转座子整合稳定细胞株
▏ 名字定义
转座因子(Transposable element,简称TE),又称“跳跃基因”,是指从基因组的一个位置移动到另一个位置的DNA序列。这类因子最早由遗传学家芭芭拉·麦克林托克于50多年前发现。起初,生物学家对麦克林托克的发现持怀疑态度。然而,在接下来的几十年里,人们逐渐发现,TE不仅会“跳跃”,而且几乎存在于所有生物体(包括原核生物和真核生物)中,而且通常数量巨大。例如,TE约占人类基因组的50%,占玉米基因组的90%。
▏ 转座子类型
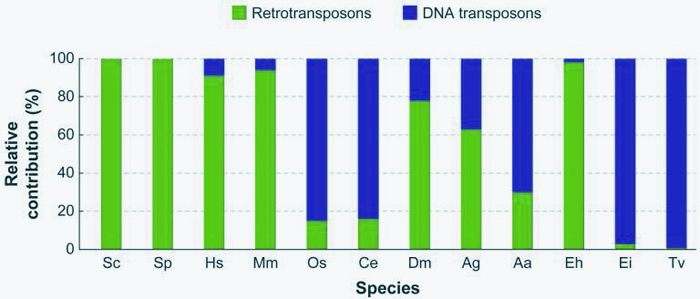
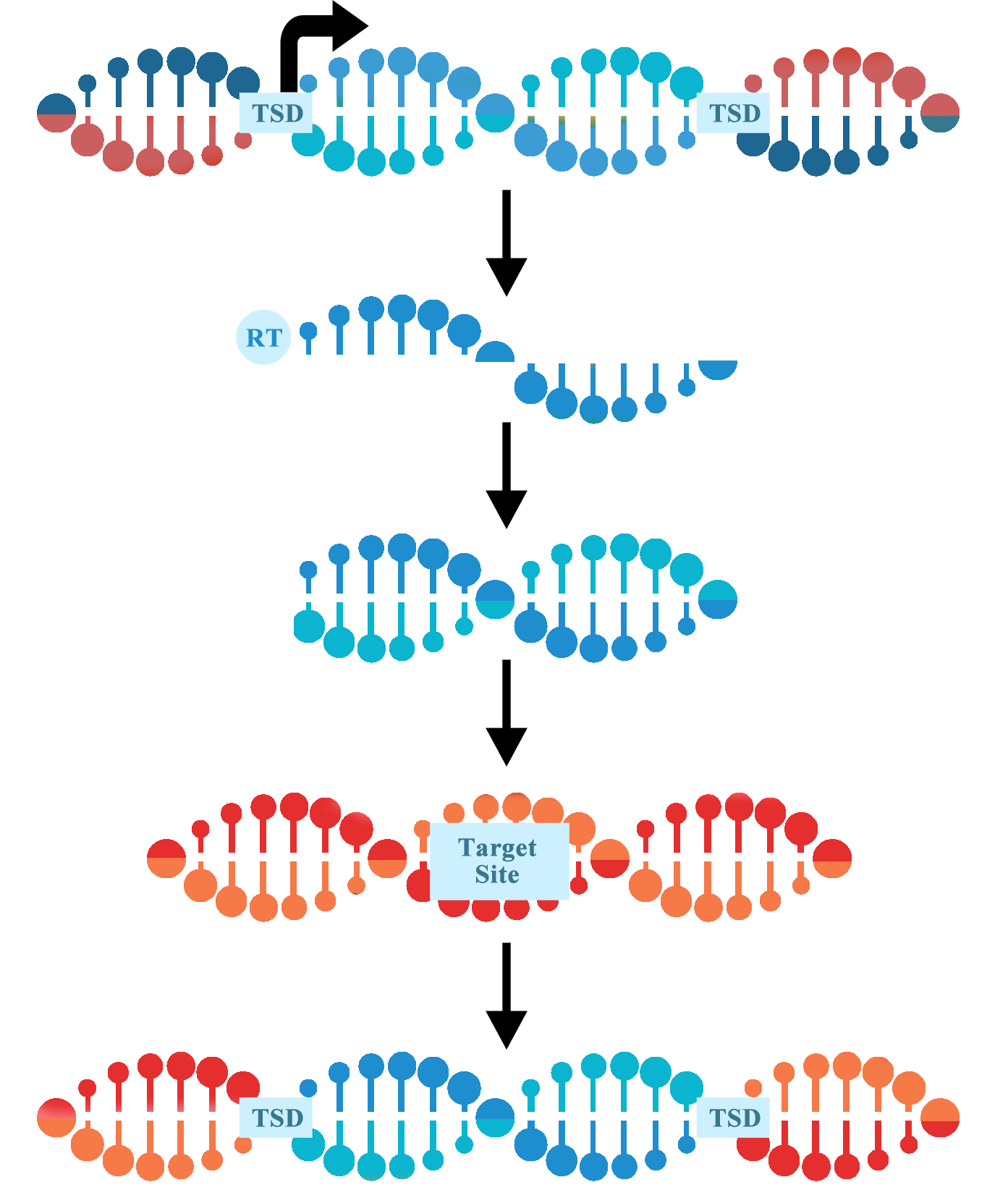
图 1:逆转录转座子转座概述。逆转录转座子通过“复制粘贴Ctrl C+V”机制进行移动。
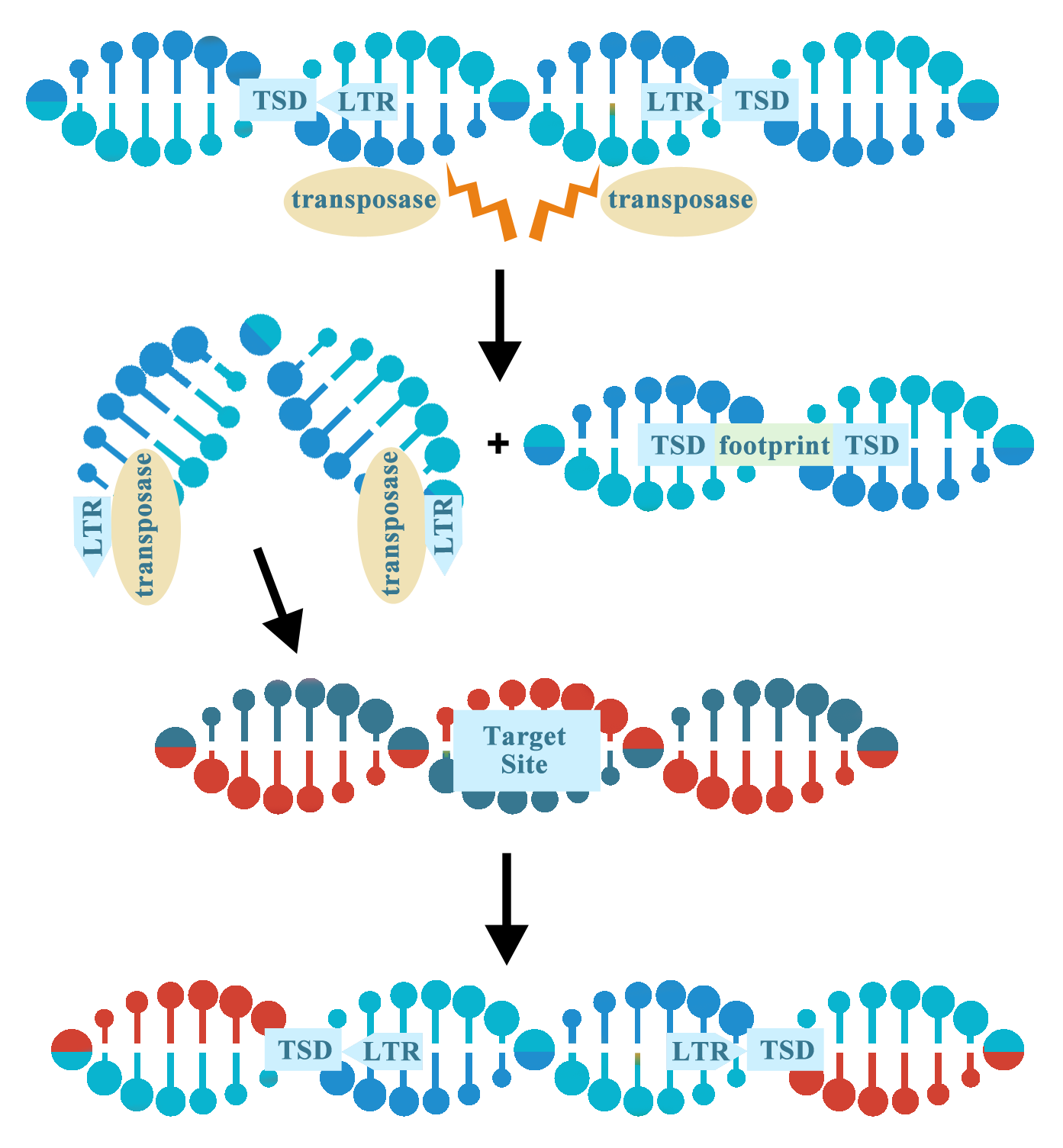
▏ 实验室中常用的DNA转座子
虽然转座子有很多不同的类型,但DNA转座子在实验室中用于基因组操作最为常见。在实验室中使用转座子时,转座酶基因以反式形式提供,以便将目标基因插入转座子的长末端重复序列(LTR)之间,类似于病毒载体的包装过程。
常见的三种适合用作研究工具的转座子系统:睡美人Sleeping beauty、PiggyBac和Tol2。
1.睡美人Sleeping beauty,简称SB
睡美人是一种合成的转座因子,由鱼类中发现的mariner。睡美人首选的整合靶位是TA二核苷酸,被转座酶切割后,它会在切除位点的末端序列上留下CAG DNA足迹。它的货物容量>100 kB,但整合效率会随着货物大小而降低。睡美人通过哺乳动物基因组进行整合,其整合特征接近随机。SB在脊椎动物中很活跃,在人类细胞中的整合速度与逆转录病毒载体相似。SB转座酶的高活性版本SB100X的效率比第一代SB转座酶高~100 倍。 hySB100x在SB100X的基础上进行了改进,转座活性提高了 30%。
2.piggyBac
piggyBac是在菜青虫中发现的 ,它的靶位是 TTAA,与其他转座子不同,它在切除后不会留下 DNA 足迹序列。piggyBac可以装载超过100 kB的DNA,并且在酵母、植物、昆虫和哺乳动物细胞(包括人类细胞)中体外和体内均有活性。piggyBac倾向于在转录起始位点、CpG 岛和 DNaseI 高敏位点进行整合。与睡美人一样,piggyBac在人类细胞中的整合效率与逆转录病毒载体相似。与密码子优化的野生型piggyBac转座酶相比,高活性PB转座酶 (hyPB)在哺乳动物细胞中的活性高约10倍。
3.Tol2
Tol2是第一个在脊椎动物中报道的活性 DNA 转座子。它是在日本青鳉鱼中发现的,因为它插入到青鳉鱼的酪氨酸酶基因中导致了白化病。与睡美人(Sleeping Beauty)和piggyBac不同,Tol2 的首选整合位点 TNA(C/G)TTATAA(G/C)TNA 具有较弱的共识序列。Tol2 可以将10-11 kB的DNA 递送到哺乳动物细胞中而不会降低效率,最大载量约为200 kB。与piggyBac类似,Tol2也倾向于在转录起始位点、CpG 岛和 DNaseI 高敏位点进行整合。Tol2 仅在脊椎动物中活跃,在人类细胞中的整合效率低于piggyBac和睡美人。Minimal Tol2或miniTol2是原始Tol2的截短版本,其转座活性提高了约 3 倍。
|
|
Sleeping Beauty (SB) |
piggyBac (PB) |
Tol2 |
|
Species of origin |
Salmonid fish |
Cabbage looper moth |
Medaka fish |
|
Classification |
Tc1/mariner superfamily |
PB superfamily |
hAT superfamily |
|
Transposable element |
~1.6 kb long |
~2.5 kb long |
~4.7 kb long |
|
Terminal regions |
IR/DRs of ~ 230 bp |
35–63 bp with outer TIRs and inner subterminal IRs |
150–200 bp containing the TIRs and subterminal regions |
|
Transposase |
340 aa |
594 aa |
649 aa (most active isoform) |
|
Footprint |
CAG |
None |
Variable |
|
Target site preference |
TA |
TTAA |
Weak consensus sequence |
|
TNA(C/G)TTATAA(G/C)TNA |
|||
|
Target site duplication |
TA |
TTAA |
8 bp |
|
Activity in species |
Various vertebrates |
Vertebrates, insects, plants, yeast |
Various vertebrates |
|
Efficiency in human cells |
Comparable to retroviral vectors |
Comparable to retroviral vectors |
Lower than PB and SB |
|
Cargo capacity |
>100 kb |
>100 kb |
>100 kb |
|
Overproduction inhibition |
Yes |
To some extent |
Lower than PB and SB |
|
Integration profile |
Close-to-random |
Biased towards TSSs, CpG islands and DNaseI hypersensitivity sites |
Biased towards TSSs, CpG islands and DNaseI hypersensitivity sites |
|
Most common parental plasmid |
pT2 |
pXL-BacII |
pTol2, miniTol2 |
|
Most hyperactive transposase |
hySB100X |
hyPB |
hTol2-M |
|
Vectors for transposon delivery |
Plasmid DNA, pFAR, MC, |
Plasmid DNA, dbDNA, |
Plasmid DNA |
|
non-integrative viral vectors, nanoparticles |
non-integrative viral vectors, nanoparticles |
||
|
Vectors for transposase delivery |
Plasmid DNA, mRNA, SNIM RNA, |
Plasmid DNA, mRNA, |
Plasmid DNA, mRNA, |
|
recombinant protein (hsSB), non-integrative viral vectors, |
non-integrative viral vectors, |
recombinant protein (His-Tol2) |
|
|
nanoparticles |
nanoparticles |
|
|
|
Clinical trials |
Yes |
Yes |
No |
▏ 常见应用
1.转座子诱变筛选:
转座子本质上是一种致突变元件,这使得它们成为检测功能丧失或功能获得突变的诱变筛选的绝佳工具。在这些筛选中,转座子编码报告基因、诱变盒或条形码。当转座子被递送到细胞或模型生物中时,它们会插入宿主的基因组中。然后,使用下一代测序检测转座子插入位点,并进行分析以确定在实验过程中哪些插入是阳性或阴性选择的。
2.转基因动物:
转基因动物通常是通过将DNA直接注射到受精卵的原核中产生的,这会导致该序列随机地整合到受精卵的基因组中,这个过程高度难以预测。然而,转座子在注射到受精卵的细胞质中后,能够有效地整合到受精卵的基因组中,而当DNA直接注射时,这个过程效率很低。睡美人、 piggyBac和Tol2都已被用于生成转基因动物,包括斑马鱼、小鼠、大鼠和兔子。
3.稳定细胞株构建
转座子是慢病毒载体构建稳定细胞株的替代方案,例如用于iPSC 重编程以及基因和细胞治疗,并且有可能克服病毒的一些局限性。TE 的有效载荷很大,使用Sleeping Beauty和piggyBac时可达100 kB,这比病毒载体有很大优势(AAV 的有效载荷约为 5 kB,慢病毒的有效载荷约为 8 kB)。转座子也比病毒载体更不容易诱导免疫反应,并且也更容易和更便宜地生成。两种传递方法都可能发生因整合而导致的基因破坏,但由于 TE 主要插入基因间区域,因此基因破坏不太令人担忧。
4.SgRNA引导的定点转座
可以借助于sgRNA来引导定点位置的转座,有报道称,INTEGRATE系统(引导 RNA 辅助靶向插入转座因子)可在细菌中实现高达10 kB大小的 DNA 的约 100% 整合。
▏ 稳定细胞株构建服务
科佰生物提供“转座子整合系统”稳定细胞株构建服务,利用该系统,我们已经成功构建超过400株稳定细胞株的模型,有着丰富的经验,欢迎沟通咨询。
|
|
质粒转染 |
病毒感染 |
Flp-FRT系统 |
转座子系统 |
基因编辑 |
||
|
脂质体 |
电转 |
慢病毒 |
|||||
|
适用细胞 |
部分细胞 |
几乎所有细胞 |
几乎所有细胞 |
内部4种细胞 |
几乎所有细胞 |
部分细胞 |
|
|
转染/感染效率 |
低 |
中 |
高 |
高 |
高 |
低 |
|
|
随机整合 |
是 |
是 |
是 |
否 |
- |
否 |
|
|
生物安全等级 |
BL1 |
BL1 |
BL2 |
BL1 |
BL1 |
BL1 |
|
|
基因装载能力 |
- |
- |
<4-5k |
- |
- |
- |
|
|
阳性率 |
低 |
高 |
高 |
高 |
高 |
低 |
|
|
周期 |
基因合成 |
2-3周 |
2-3周 |
2-3周 |
2-3周 |
2-3周 |
2-3周 |
|
病毒包装 |
- |
- |
1周 |
- |
- |
- |
|
|
混合克隆筛选 |
1-2周 |
1-2周 |
1-2周 |
1-2周 |
1-2周 |
1-2周 |
|
|
单克隆筛选 |
>3 周 |
>3 周 |
>3 周 |
可选 |
可选 |
>3 周 |
|
|
合计 |
> 8-10 周 |
> 8-10 周 |
>9-11周 |
5-7 周 |
5-7 周 |
> 8-10 周 |
|
|
工作负荷 |
高 |
中 |
中 |
低 |
低 |
高 |
|


