慢病毒整合位点标准品的应用现状与创新
发布时间:2025/07/29分类:技术文章来源:科佰生物
在 CAR-T 细胞疗法等基因治疗领域,慢病毒载体的整合位点检测是保障临床安全的关键环节。慢病毒载体在将治疗性基因导入细胞时,可能随机整合到宿主基因组中,存在插入突变风险,若整合到原癌基因附近,可能诱发细胞恶性转化;若整合到抑癌基因内,可能导致其功能失活。因此,FDA、CDE 等监管机构明确要求对慢病毒整合位点进行精准检测与风险评估。
慢病毒整合位点检测方法
目前检测慢病毒整合位点的主要平台为高深度测序(NGS)。
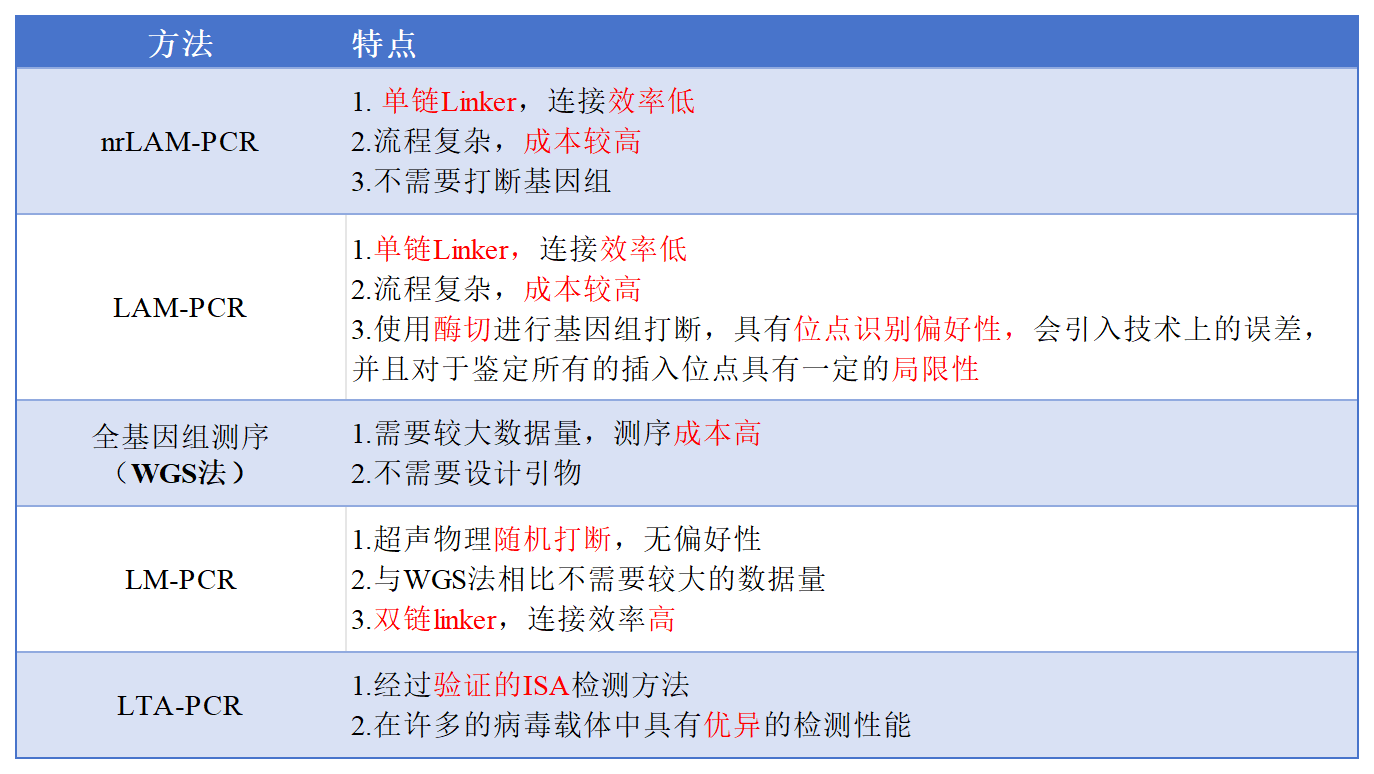
Table 1. 主流的慢病毒整合位点检测方法比较
标准化的整合位点标准品不仅能校准检测方法的灵敏度与准确性,还能为不同实验室的数据比对、方法验证提供“通用标尺”。在进行方法学开发、性能验证、试剂盒开发、日常检测质控中都需要使用标准品。
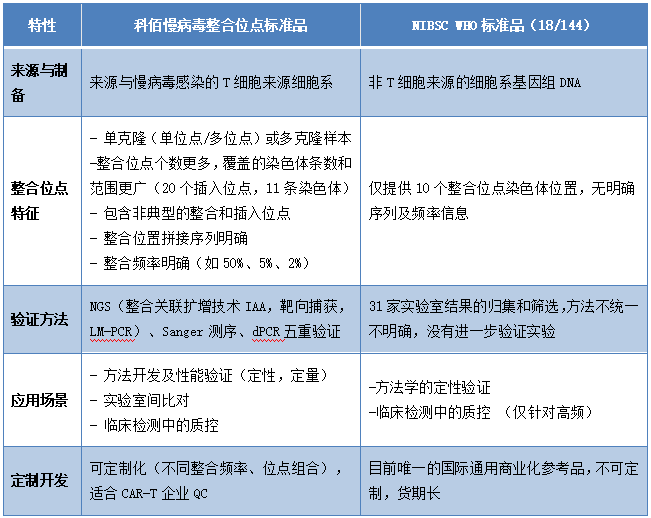
NIBSC WHO标准品(18/144)
目前商业化常见的慢病毒整合位点标准品为NIBSC WHO 标准品(18/144),它由NIBSC制备并发起,联合全球13个国家的31家实验室检测的结果,确定该标准品包含共计10个慢病毒已知整合位点,分布在10条不同的染色体上,该产品面世以来已被众多国内外检测机构用于慢病毒整合位点检测项目的性能验证及临床检测应用中的质控。尽管如此该产品在实际应用过程中仍存在一些短板和痛点,其中包括:
1
该产品选取公布的位点信息仅来自于31家实验室大多数获得的较为一致的结果,不排除有一些潜在的未被公布,对一些复杂的非典型的整合和插入方式(例如一端或部分LTR发生缺失的整合位点)可能存在漏报;
2
该产品的参数来自于多家检测机构检测结果的综合和筛选,方法学上并未明确和统一,也并未就相应的位点进一步进行验证性实验;
3
该产品只公布了整合位点的染色体位置,而没有明确插入位置两端的确切拼接序列,没有完整精确呈现各位点的插入情况;
4
该产品并未提供整合位点的位点频率数据,只能作为定性参考品使用,不能直接用于准确性,精密度,检测下限等相关性能验证;
5
该产品经我们验证是来自于一个单克隆的核酸样本,而目前临床上需要检测整合位点的CAR-T样本多为多克隆样本。单克隆与多克隆样本相比存在整合位点较为单一,位点频率较高容易检出等特点,因此对应临床上大量多克隆样品,整合位点繁多且低频的特点,该产品并不能很好的模拟实际样本的检测。
科佰生物自主开发
针对上述痛点和缺陷,科佰生物自主开发了新一代的慢病毒整合位点标准品,该产品通过整合关联扩增技术(IAA)以及常用的靶向捕获技术和LAM-PCR三种方式结合NGS发现并验证标准品的整合位点,最大程度地保证了标准品中所有整合位点的准确完整报道,进一步结合了一代(Sanger)测序和数字PCR,精确定性和定量了所有整合位点的序列及频率信息,做到了五重验证。
另外为了进一步满足CAR-T细胞整合位点的临床检测需求,模拟临床的实际情况。我们采用了T细胞来源的细胞系制备该标准品,并且除了单克隆样品,还制备了特定低频(位点频率低至2%)的多克隆样本,且包含位点更多,覆盖的染色体条数和区域更广,可直接用于检测方法的性能验证,确定方法精密度,准确性,检测限等指标,也更贴合临床检测中的实际情况。
两种标准品的对比分析
科佰生物产品特点
1
高度模拟临床样本
T细胞来源系作为宿主,更贴近CAR-T治疗的实际场景。通过单克隆+多克隆标准品的设置,模拟随机整合和多克隆多位点低频的复杂性。
2
多重技术验证体系
五步正交验证(NGS(整合关联扩增技术(IAA,靶向捕获, LAM-PCR)+ Sanger确认 + dPCR定量),确保位点序列与频率的准确性。在验证LTR整合基因组位点的基础上,进一步验证了载体上其他基因序列在基因组上的非典型整合位点。
3
灵活的频率梯度设计
可提供 5%和2%低频整合样本,满足FDA对插入突变风险评估的灵敏度需求。
4
标准化与可扩展性
单克隆样本可作为“标准模块”,按需混合生成多克隆标准品,适应不同检测需求。
慢病毒整合位点标准品
针对这一要求,科佰生物推出多款慢病毒整合位点标准品。
| 货号 | 名称 |
|
Lentiviral Vector Integration Site Reference Standard |
|
| CBPL0003 |
Lentiviral Vector Integration Site Reference Standard II (Single site) |
| CBPL0005 |
慢病毒整合位点分析参考试剂IV (含有性染色体插入) |
| CBPL0006 |
慢病毒整合位点分析阴性参考试剂 |
| CBPL0007 |
慢病毒多克隆多整合位点参考品-5% |
| CBPL0008 |
慢病毒多克隆多整合位点参考品-2% |
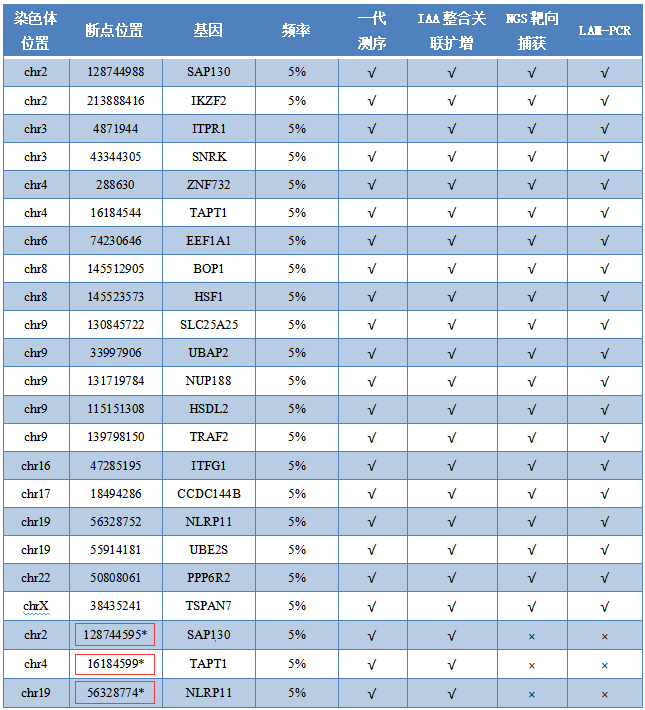
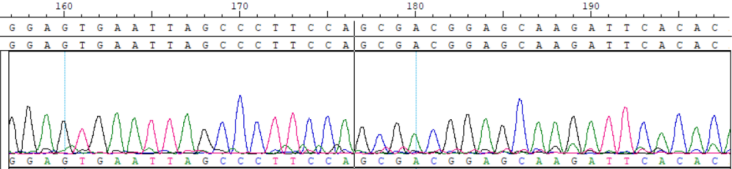
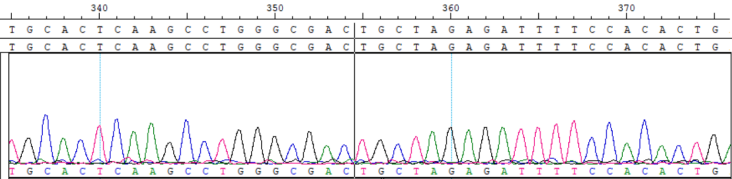
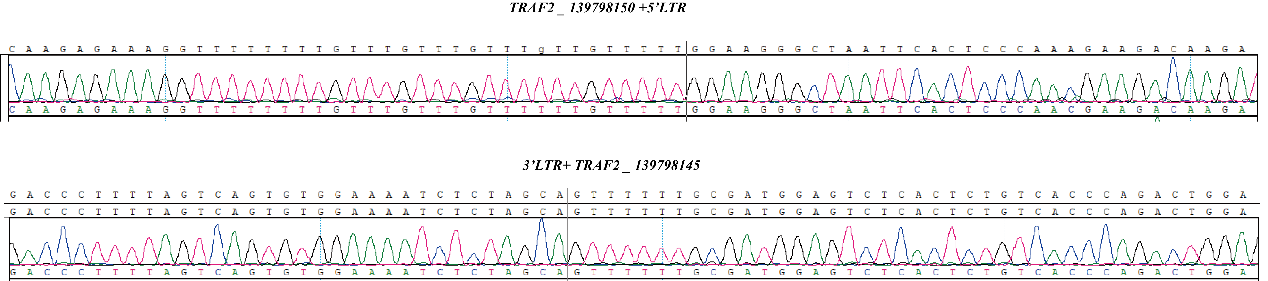
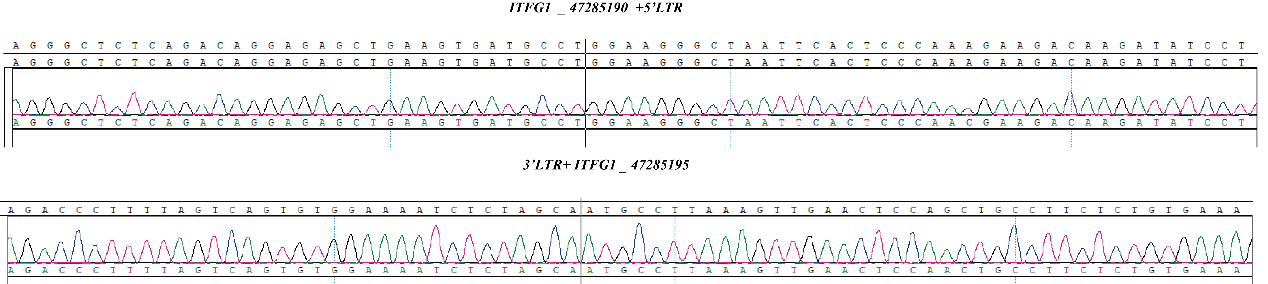
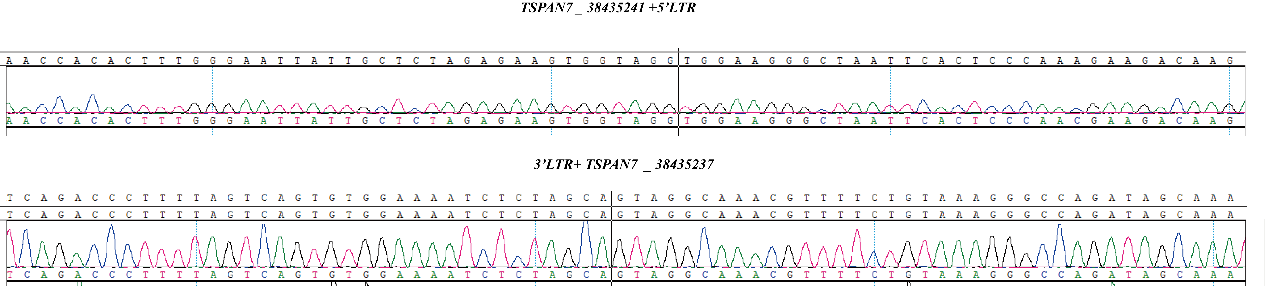
Fig1.CBPL0007 慢病毒多克隆多整合位点参考品-5%五步正交验证结果
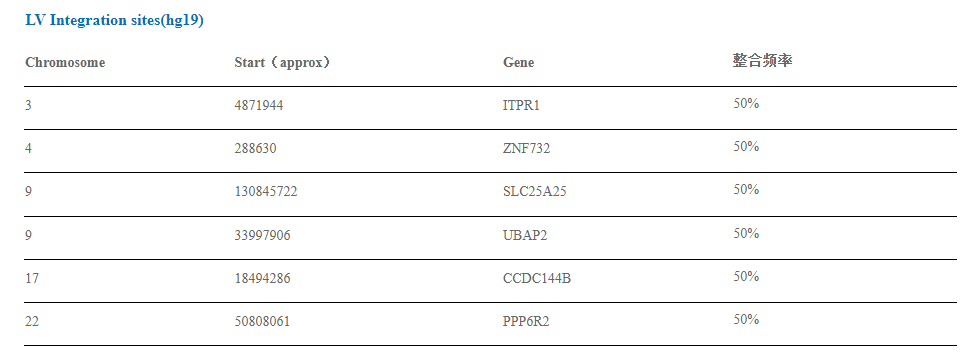
Fig 2. Results of CBPL0002 by NGS (Probe capture method).
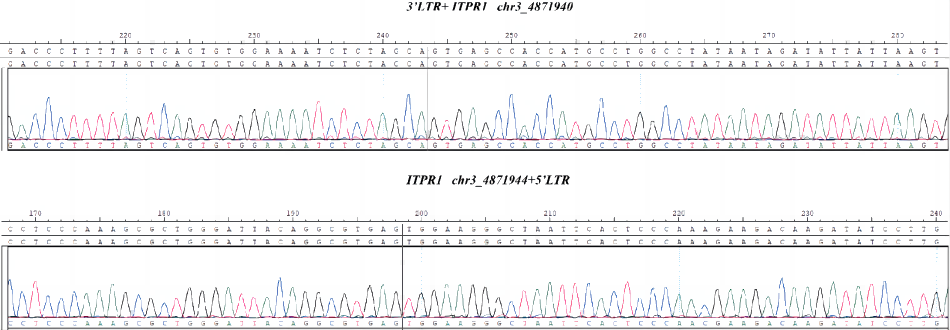
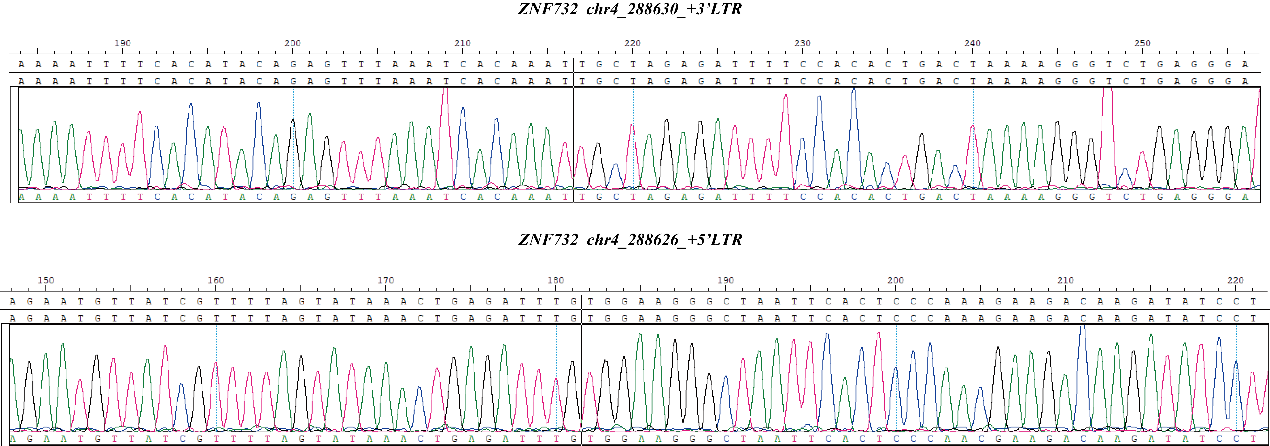
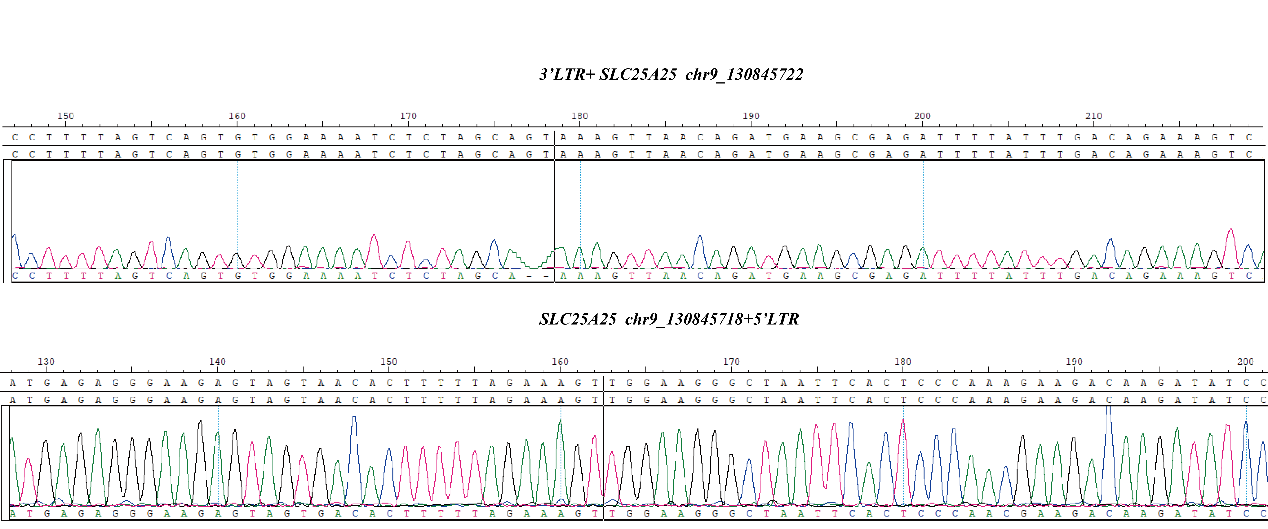
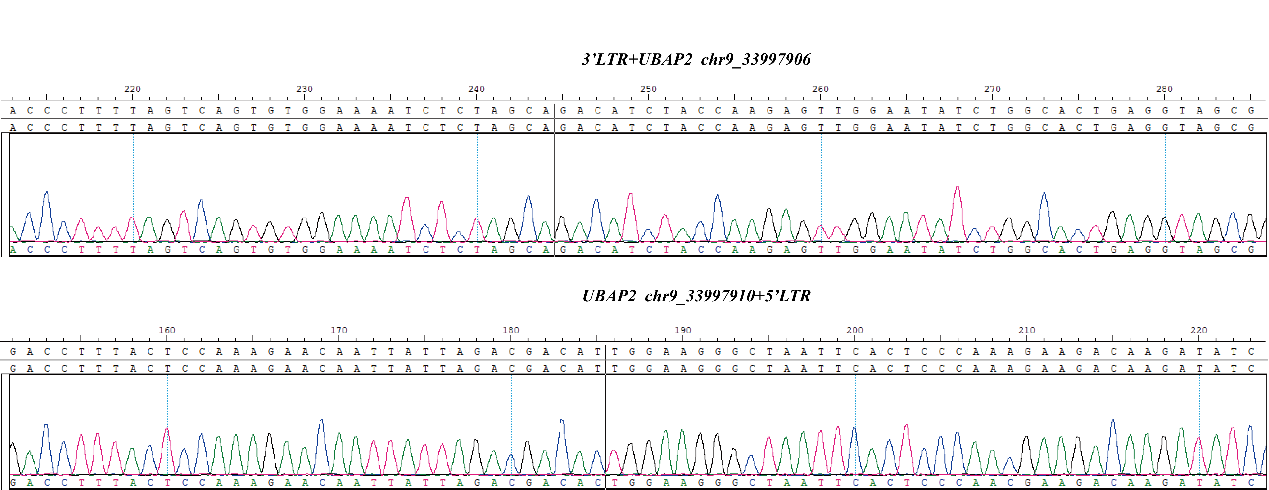
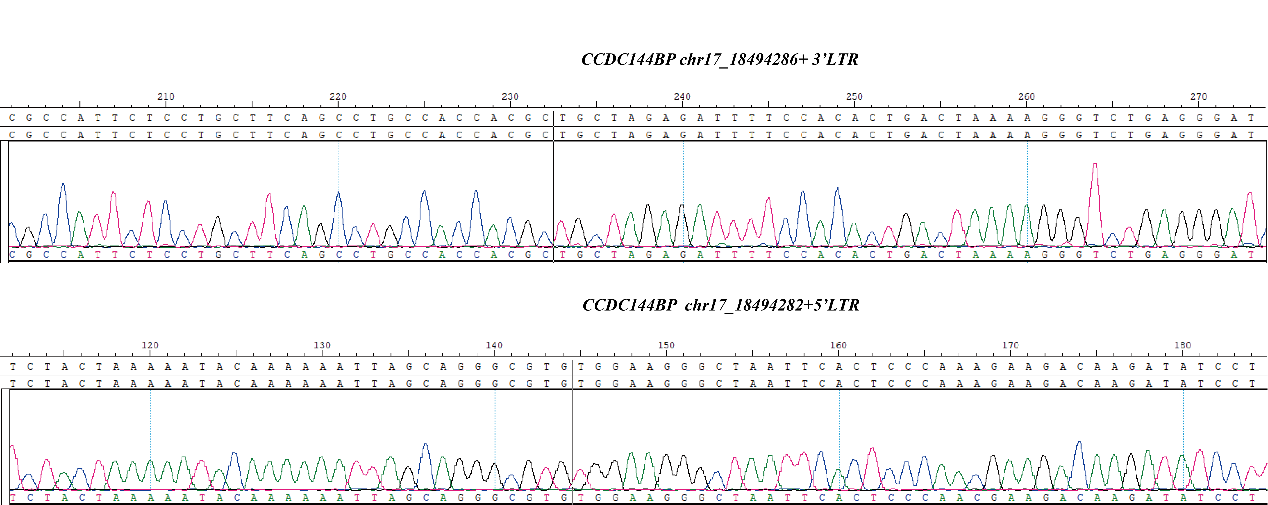
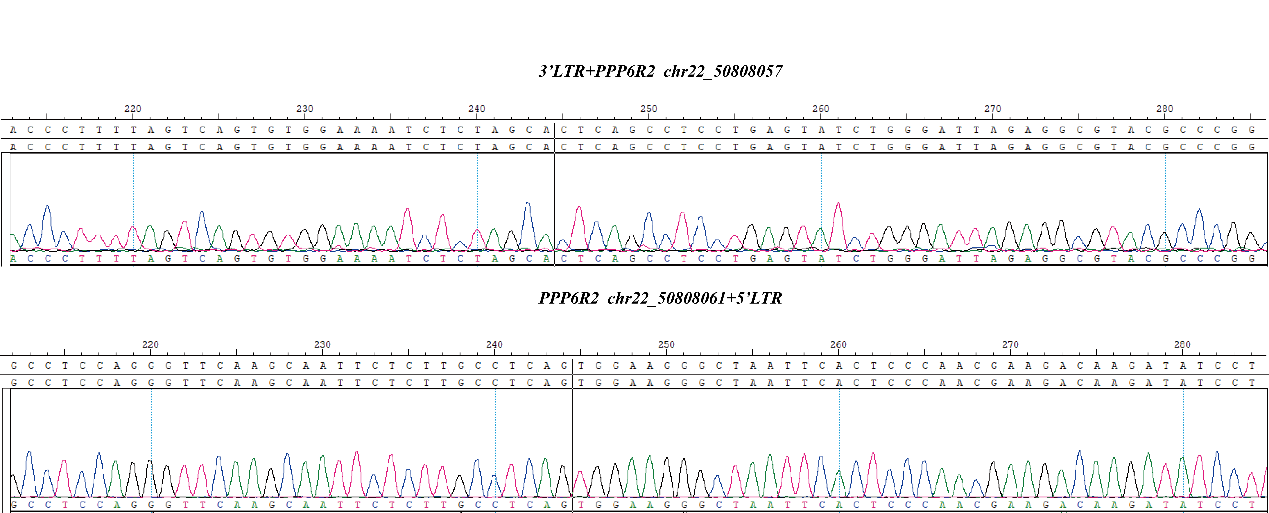
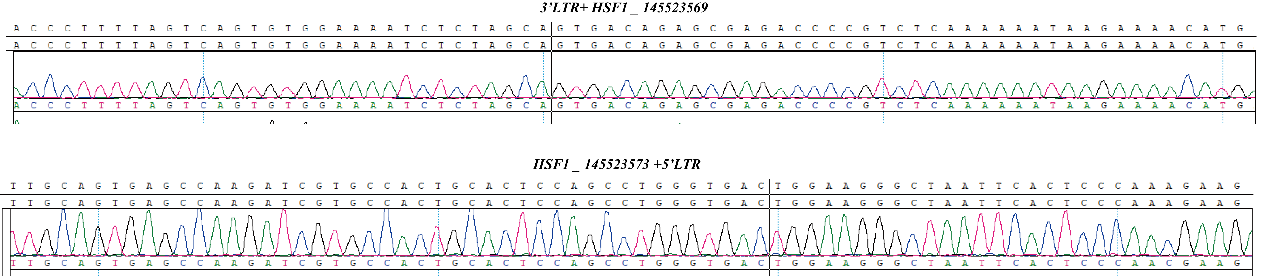
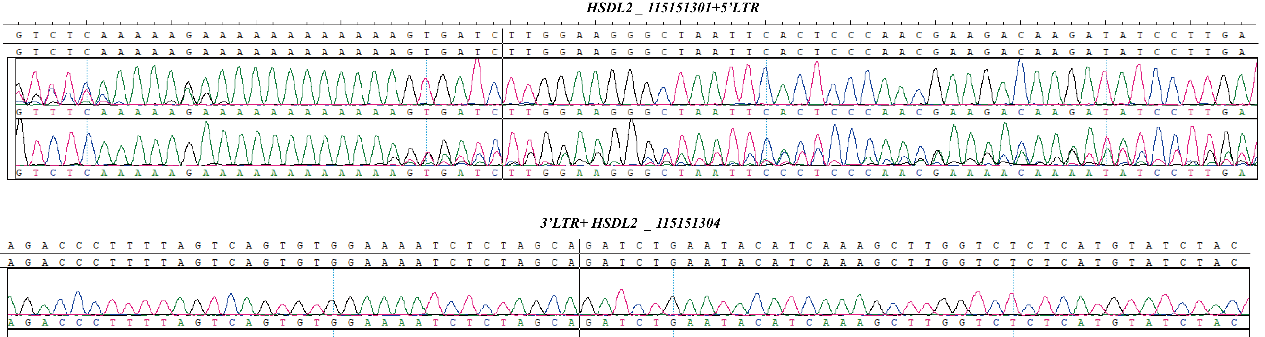
Fig 3.Results of CBPL0002 by Sanger sequencing.
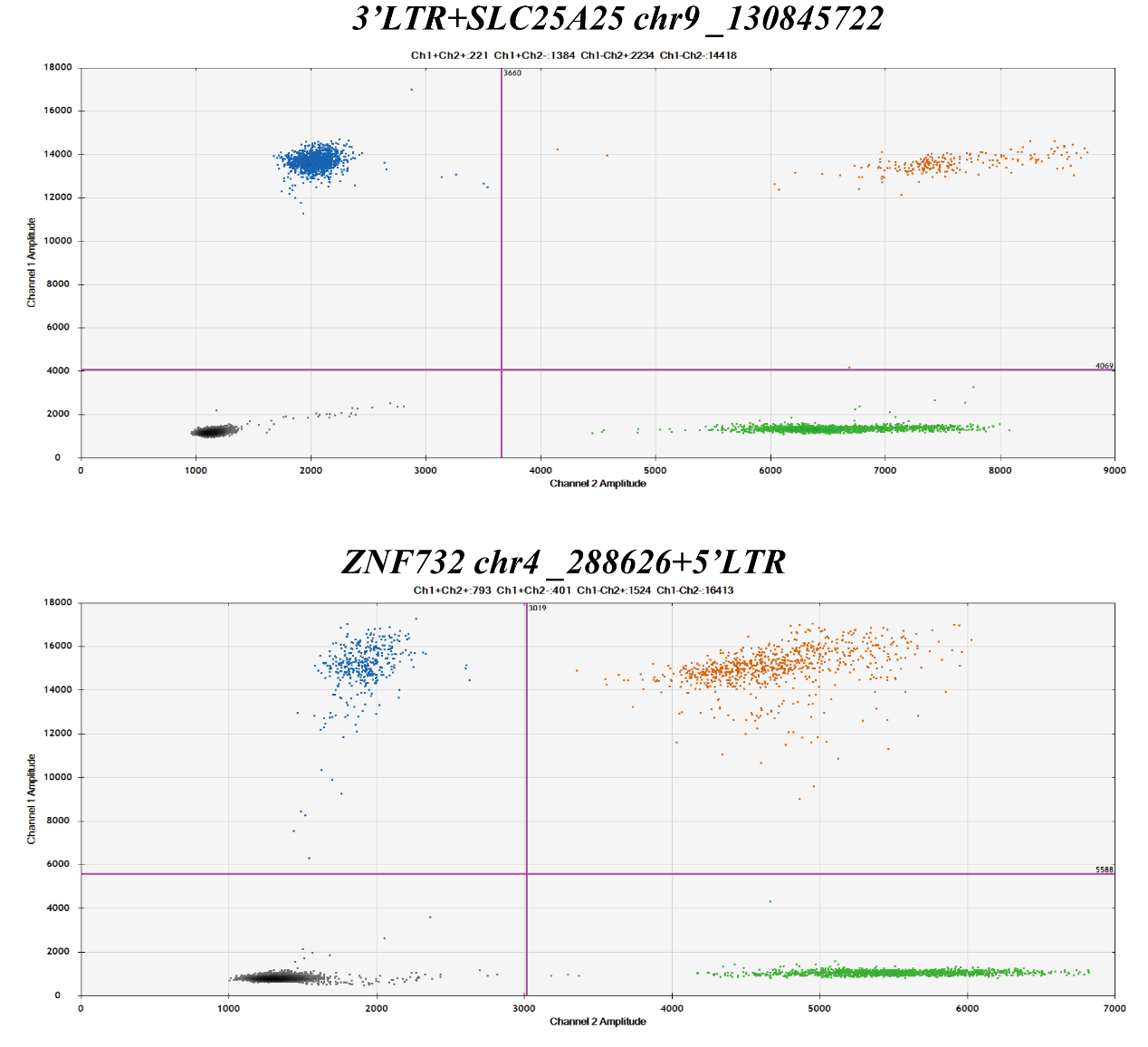
Fig 4.Results of CBPL0002 by ddPCR.